vietnam medical journal n01 - MARCH - 2020
178
đồng tử giãn liệt. Các đánh giá về độ mở góc
không thấy khác biệt giữa các hình thái đóng góc
NĐT và MMP. Dấu hiệu quan trọng có giá trị
chẩn đoán hình thái là dấu hiệu lạc đà 2 bướu
với độ đặc hiệu trên 90%.
TÀI LIỆU THAM KHẢO
1. Nguyễn Thị Thu Hiền (2012). Ứng dụng máy
siêu âm sinh hiển vi đánh giá sự thay đổi bán phần
trước nhãn cầu sau laser cắt mống mắt chu biên
điều trị dự phòng Glôcôm góc đóng nguyên phát,
Luận văn tốt nghiệp bác sĩ nội trú.
2. Nguyễn Văn Độ, Phạm Thị Thu Thy (2016).
Kết quả lâu dài phương pháp tạo hình góc tiền
phòng bằng laser trong điều trị glôcôm góc đóng
nguyên phát, Luận Văn Thạc Sỹ, Trường Đại Học Y
Hà Nội
3. Chelvin C.A, Sng MBBChir, FRCS(Ed), et al.
(2014). Pretreatment Anterior Segment Imaging
During Acute Primary Angle Closure:Insights into
Angle Closure Mechanisms in the Acute Phase.
American Academy of Ophthalmology. 21:119-125.
4. Paul J Foster.FRCSEd, Jamyanjav Baasanhu.
MD, Poul Helge Alsbirk. MD, et al. (1996).
Glaucoma in Mongolia: a population-based survey
in Hövsgöl Province, northern Mongolia. Archives
of ophthalmology. 114(10):1235-1241.
5. Yi-an You. MD, Le-ru Zhu. MD, Ji-quan Wen.
MD, et al. (2015). Ultrasound Biomicroscopic
Evaluation of Uveal Effusion in Acute Primary Angle
Closure. J Glaucoma. 24:656–661.
6. Jian-Gang Yang, Jian-Jun Li, Hua Tian, et al.
(2017). Uveal effusion following acute primary
angle-closure: aretrospective case series. Int J
Ophthalmol .10(3):406-412.
7. Gazzard G, Friedman DS, Devereux JG, et al.
(2003). A prospective ultrasound biomicroscopy
evaluation of changes in anterior segment
morphology after laser iridotomy in Asian eyes.
Ophthalmology. 110(3): 630-8.
8. Kiuchi Y, Kanamoto T, Nakamura T (2009).
Double hump sign in indentation gonioscopy is
correlated with presence of plateau iris
configuration regardless of patent iridotomy. J
Glaucoma.18(2):161-4.
XÂY DỰNG KỸ THUẬT MULITPLEX PCR KHUẾCH ĐẠI 14 CHỈ THỊ STR
ỨNG DỤNG TRONG PHÂN TÍCH DI TRUYỀN TRƯỚC CHUYỂN PHÔI
BỆNH HEMOPHILIA A
Đng Tiến Trường*, Nguyn Trường Giang*, Nguyn Duy Bắc*
TÓM TẮT45
Mục tiêu: Xây dựng kỹ thuật Mulitiplex-PCR, ứng
dụng trong PGT-M bệnh Hemophilia A. Phương pháp
nghiên cứu: Sử dụng phần mềm tin sinh xác định Tm;
Tối ưu hóa nhiệt độ gắn mồi bằng phản ứng PCR đơn;
Tối ưu nồng độ primer của phản ứng Multiplex PCR dựa
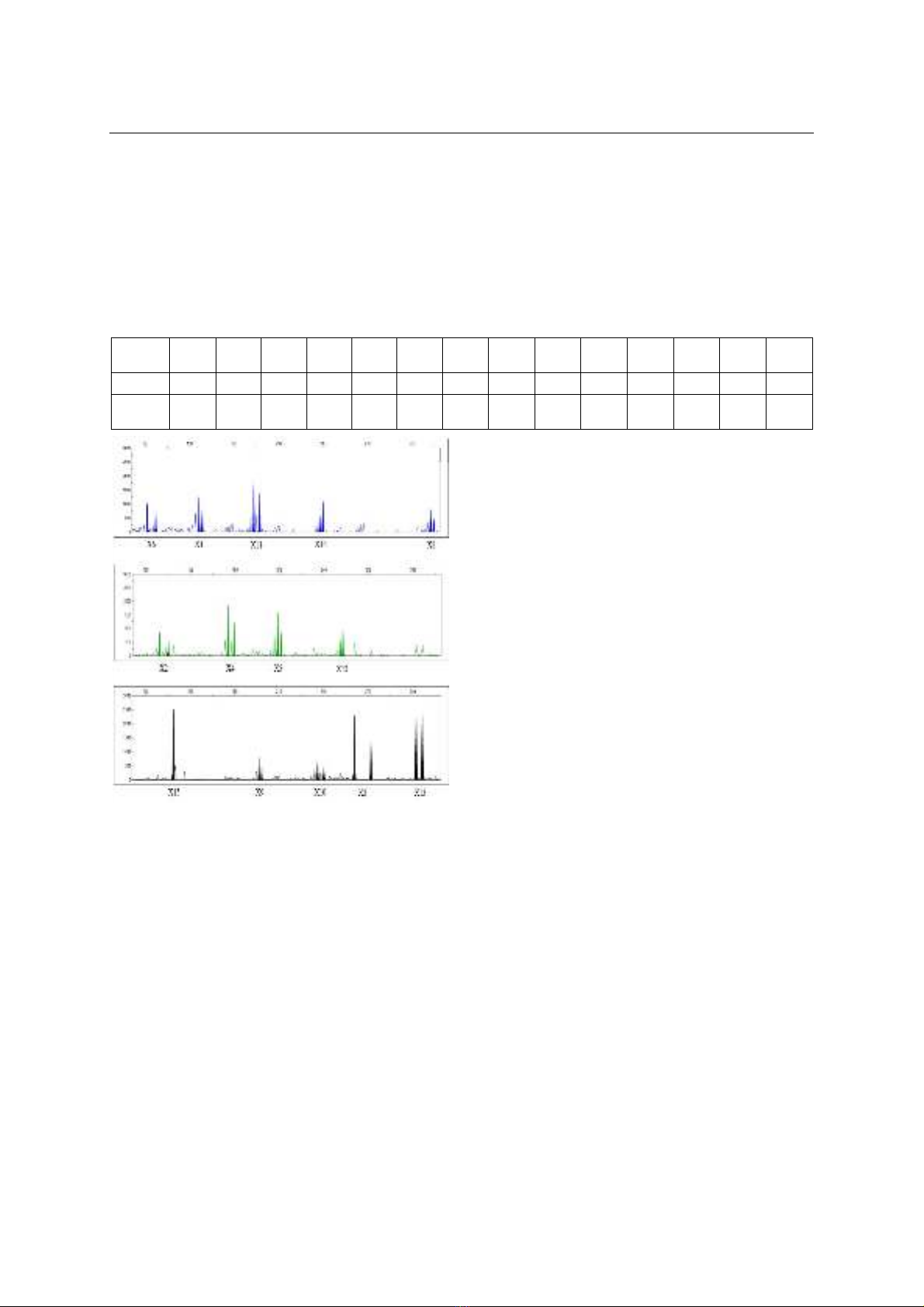
trên tín hiệu trên kết quả điện di mao quản. Kết quả
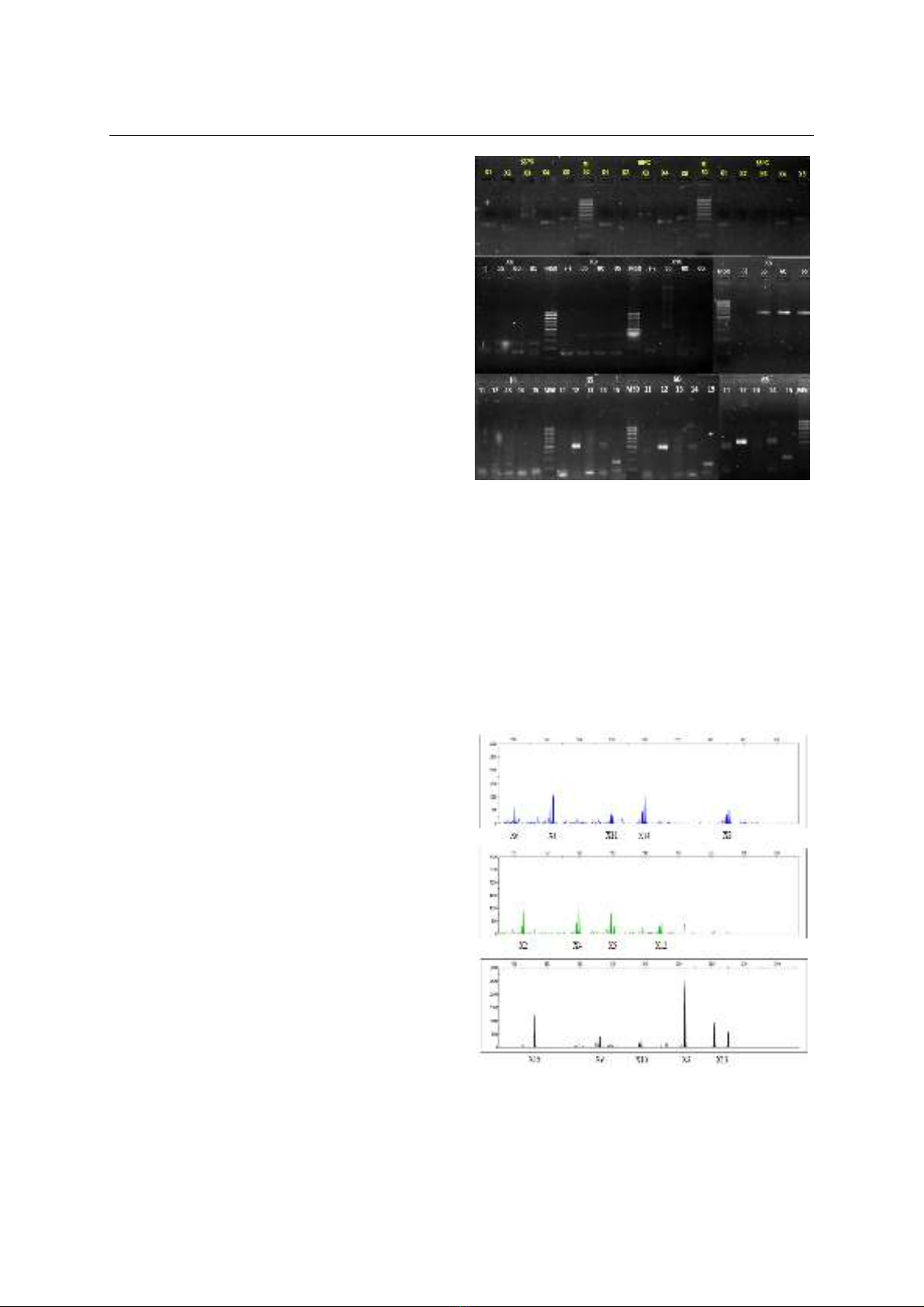
và kết luận: Xây dựng và tối ưu hóa được kỹ thuật
Mutiplex PCR khuếch đại đồng thời 14 cặp chỉ thị STR
với nồng độ các cặp primer từ 0,15-0,7µM, ở Ta là
60oC. Quy trình được thiết lập đáp ứng yêu cầu phân
tích di truyền trước chuyển phôi bệnh Hemophilia A.
Từ khóa:
Multiplex PCR, chỉ thị STR, Hemophilia A.
SUMMARY
ESTABLISHMENT OF MULTIPLEX-PCR OF
14 STR MARKERS FOR PGT-M HEMOPHILIA A
Objective: Establish Multiplex PCR reaction which
is applied for preimplantation genetic testing for the
monogenic disorder (PGT-M) in Hemophilia A.
Method: Using bioinformatics software to determine
Tm; Optimize Ta by single PCR reaction; Optimize the
primer concentration of Multiplex-PCR based on the
*Học viện Quân y
Chịu trách nhiệm chính: Nguyễn Duy Bắc
Email: bac_hvqy@yahoo.com
Ngày nhận bài: 3.01.2020
Ngày phản biện khoa học: 20.2.2020
Ngày duyệt bài: 28.2.2020
signal of capillary electrophoresis results. Result and
Conclusion: A Multiplex-PCR of 14 STR markers with
primer concentration from 0.15-0.7µM, at 60oC was
set up successfully. This procedure can be applied in pre-
implantation genetic testing for PGT-M for Hemophilia A.
Key words
: Multiplex PCR, STR marker,
Hemophilia A.
I. ĐẶT VẤN ĐỀ
Hemophilia A là bệnh rối loạn đông máu di
truyền. Nguyên nhân do tổn thương gen F8 trên
nhiễm sắc X (Xq28), giảm sản xuất yếu tố VIII,
gây chảy máu không cầm. Tỷ lệ mắc bệnh
khoảng 1/5000 người nam[1]. Điều trị hemophilia
A tốn kém nhưng không giải quyết được nguyên
nhân bị bệnh. Vì vậy, dự phòng không sinh con bị
bệnh hoặc không mang gen bệnh là rất cần thiết.
Gen gây bệnh Hemophilia A rất lớn. Đến nay,
có hơn 2.000 đột biến của gen F8 được xác định.
Trong đó, đột biến đảo đoạn intron 22 và intron
1 chiếm 50% số ca bệnh nặng. Kỹ thuật phân
tích di truyền trước chuyển phôi đơn gen
(Preimplantation Genetic Testing for monogenic
Disease, PGT-M) dựa trên việc xác định trực tiếp
đột biến không thể áp dụng cho các đột biến
đảo đoạn này. Bên cạnh đó, hiện tượng khuếch
đại hỏng alen (Allele DropOut-ADO) ở mẫu tế
bào phôi, được xác định trên 40% các ca chẩn