TNU Journal of Science and Technology
230(05): 353 - 360
http://jst.tnu.edu.vn 353 Email: jst@tnu.edu.vn
ALLELES FREQUENCY OF 21 STR LOCI OF VIETNAM KINH POPULATION
Duong Thi Thu Thuy1*, Le Thi Quynh2, Tran Van Cuong2, Le Thi Thu Thuy1, Bui Anh Tuan1
1Institute of Forensic Science - Ministry of Public Security
2People’s Security Academy - Ministry of Public Security
ARTICLE INFO
ABSTRACT
Received:
29/8/2024
To draw conclusions from forensic DNA typing to trace individuals or
paternity testing, it is necessary to include statistic probability based
on allele and genotype frequencies specific to each population. The
use of 21 core STR loci in DNA typing are widely implemented,
however data on extended STR loci in the Vietnamese (Kinh)
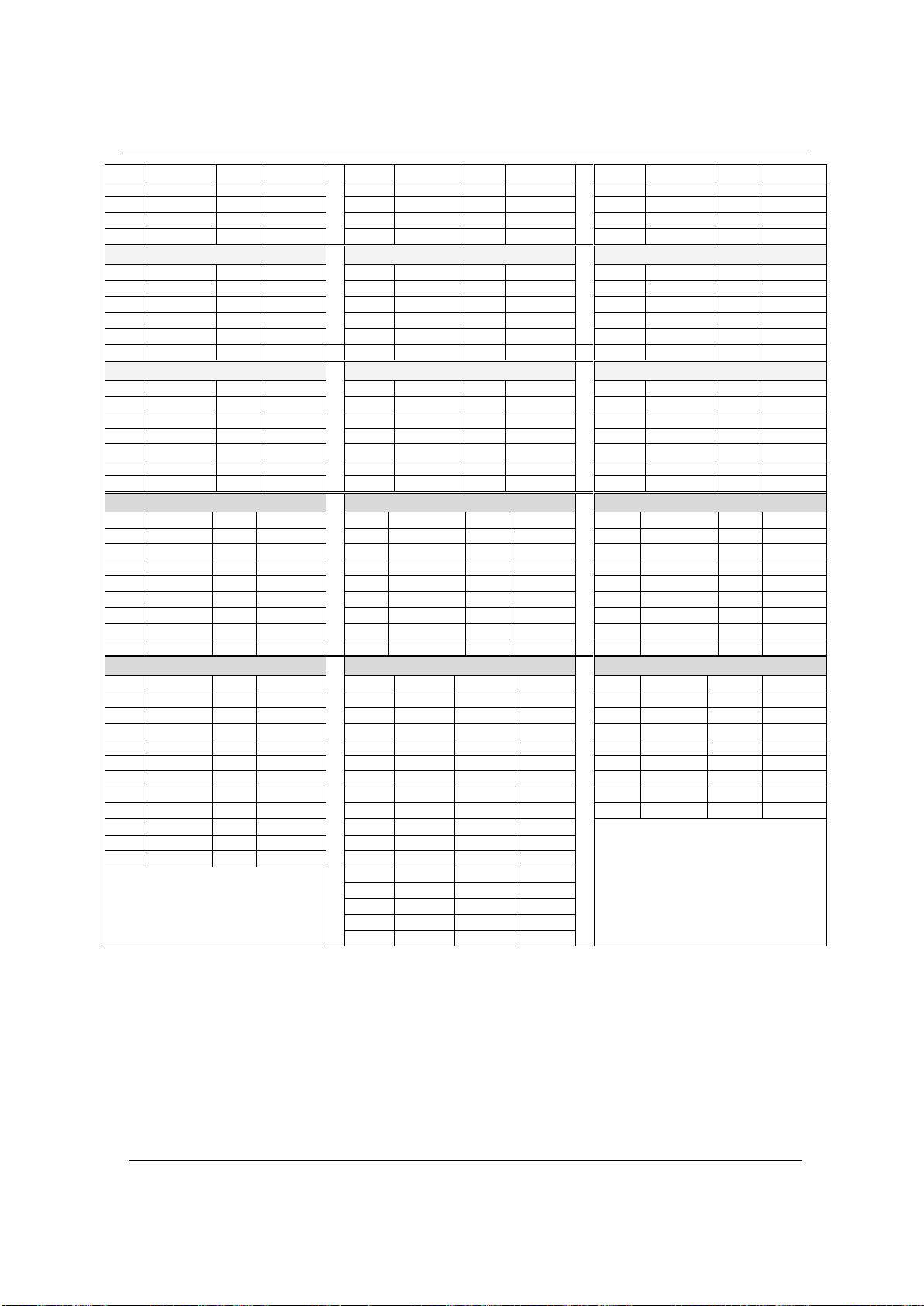
population is still lacking. In this study, the frequency and
polymorphism assessment indices of alleles in the studied population
were calculated and surveyed to build a table on the frequency of
alleles of 21 gene loci of Kinh population according to the GlobalFiler
kit. The results built an allele frequency table of 21 gene loci,
identified a number of typical genetic characteristics such as the
highest and the lowest polymorphism locus, and some common
alleles. Research results are an essential data, providing scientific
basis for each forensic DNA conclusion, serving litigation
requirements.
Revised:
06/02/2025
Published:
07/02/2025
KEYWORDS
DNA profiling
21 STR loci
GlobalFiler
Allele frequency
Vietnamese
NGHIÊN CỨU TẦN SUẤT CÁC ALEN CỦA 21 LOCUS STR
Ở NGƯỜI KINH VIỆT NAM
Dương Thị Thu Thủy1*, Lê Thị Quỳnh2, Trần Văn Cường2, Lê Thị Thu Thủy1, Bùi Anh Tuấn1
1Viện Khoa học hình sự - Bộ Công an, 2Học viện An ninh nhân dân - Bộ Công an
THÔNG TIN BÀI BÁO
TÓM TẮT
Ngày nhận bài:
29/8/2024
Để đưa ra kết luận giám định ADN phục vụ truy nguyên cá thể hoặc
xác định quan hệ huyết thống, cần kèm theo con số xác suất dựa trên
dữ liệu tần suất alen và tần suất kiểu gen đặc trưng cho mỗi quần thể.
Việc sử dụng 21 locus STR lõi trong giám định ADN hình sự đã
được thực hiện rộng rãi trên thế giới và ở Việt Nam, tuy nhiên dữ liệu
của các locus STR mở rộng trên quần thể người Việt (Kinh) hiện vẫn
còn thiếu. Trong nghiên cứu này, tần suất và các chỉ số đánh giá tính
đa hình các alen trong quần thể nghiên cứu được tính toán khảo sát
nhằm xây dựng được bảng dữ liệu về tần suất các alen của 21 locus
gen của người Việt (Kinh) theo bộ kit GlobalFiler. Kết quả nghiên
cứu đã xây dựng được bảng tần suất alen của 21 locus gen, xác định
được một số đặc điểm di truyền đặc trưng như locus có tính đa hình
cao nhất và thấp nhất, một số alen phổ biến. Kết quả nghiên cứu là
nguồn dữ liệu thiết yếu, cung cấp căn cứ khoa học cho mỗi kết luận
giám định ADN, phục vụ các yêu cầu tố tụng.
Ngày hoàn thiện:
06/02/2025
Ngày đăng:
07/02/2025
TỪ KHÓA
Giám định gen (DNA)
21 locus gen
GlobalFiler
Tần suất các alen
Người Việt Nam
DOI: https://doi.org/10.34238/tnu-jst.11032
* Corresponding author. Email: thuthuysinhhoc@gmail.com