1
MỒI PCR/ PCR PRIMER
Vũ Thị Thúy Hằng
Nội dung
1. Khái niệm
2. Nguyên tắc chung trong thiết kế mồi
3. Một số phần mềm trong thiết kế mồi
Lớp Học Phần VNUA - Khoa Nông Học - Học Viện Nông Nghiệp Việt Nam
https://sites.google.com/site/lophocphank57vnua/home

2
The Polymerase Chain Reaction (PCR) làm cuộc cách mạng trong khoa
học sự sống vì cho phép nhân bản một số lượng lớn DNA một cách
thuận tiện, hiệu quả, và có độ cậy
Được phát minh năm 1983 bởi Kary Mullis, dành giải Nobel năm 1993
Kỹ thuật PCR được sử dụng rộng rãi khi phát hiện ra Taq polymerase,
một DNA polymerase trong vi khuẩn Thermus aquaticus, bền với nhiệt.
Nguyên liệu chính dùng trong PCR:
- DNA nucleotides: thành phần hình thành nên đoạn DNA mới
- DNA khuôn: trình tự/đoạn DNA muốn nhân bản
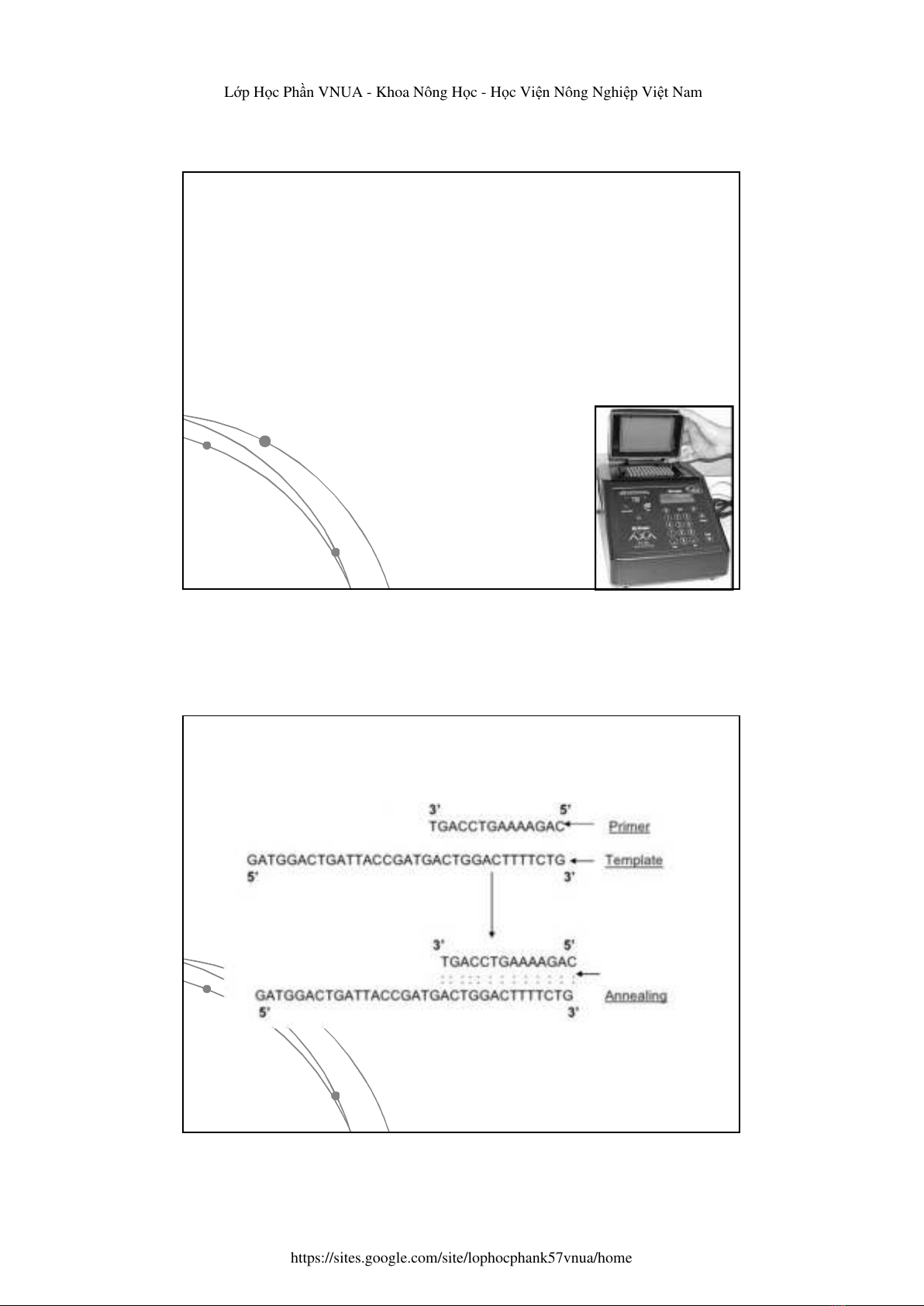
- Primers/Mồi: đoạn DNA mạch đơn gồm 16--50 nucleotide
bắt cặp bổ sung với một vùng ngắn ở 2 đầu của đoạn DNA khuôn
- DNA polymerase: enzyme bền với nhiệt độ dùng để tổng hợp đoạn
DNA mới
PCR: công nghệlàm thay đổi thếgiới
Mồi – quyếtđịnh thành công của phảnứng PCR
Đặc hiệu/
Chuyên biệt?
Gắn với DNA
khuôn
Lớp Học Phần VNUA - Khoa Nông Học - Học Viện Nông Nghiệp Việt Nam
https://sites.google.com/site/lophocphank57vnua/home
3
Mồi là đoạn nucletotit ngắn, bắt cặp bổ sung với đầu 5’ hay 3’
của DNA mạch khuôn
Thiết kế mồi là tạo ra một đoạn trình tự nucleotit ngắn sử dụng
trong nhân bản một vùng đặc hiệu của DNA. Mồi được thiết kế
dựa vào 2 vùng trình tự đã biết nằm ở 2 đầu đoạn gen cần nhân
bản
Mồi thiết kế được dùng để :
- Tìm gen mới
- Xây dựng công cụ để nhận biết, phân biệt
- Tối ưu sản phẩm PCR
1. Khái niệm
Lớp Học Phần VNUA - Khoa Nông Học - Học Viện Nông Nghiệp Việt Nam
https://sites.google.com/site/lophocphank57vnua/home

4
2. Nguyên tắc chung khi thiết kế mồi
Trước khi thiết kếmồi –
Đừng phát minh lại bánh xe!
Lớp Học Phần VNUA - Khoa Nông Học - Học Viện Nông Nghiệp Việt Nam
https://sites.google.com/site/lophocphank57vnua/home
5
Trước khi thiết kếmồi –
Tìm kiếm và sửdụng đúng nguồn thông tin!
Mồi thiết kế dùng để làm gì?
Nhân bản DNA nói chung?
Phát hiện sự khác nhau ở cấp độ từng nucleotide/SNPs?
Nghiên cứu về Methylation (nhóm methyl gắn vào và làm DNA
thay đổi chức năng)
Trong PCR để định lượng được DNA nhân bản (Real-time PCR)
Làm mồi dò cho microarray?
Cho nhiều PCR trong cùng một phản ứng (sử dụng nhiều mồi)
Bắt đầu từ đâu?
Trình tự DNA mạch đơn/ Trình tự protein?
Nhiều file trình tự DNA/protein?
Ngân hàng gen (GenBank ID/Gene ID/Gene Symbol/rsSNP ID?
Sau đó, xem xét một sốlựa chọn!
Hầu hết các mồi có thể được thiết kế từ một số phần mềm khác
nhau
Các phần mềm khác nhau có thể cho kết quả khác nhau, chủ yếu về:
Cách tiếp cận
Tiêu chí thiết kế và mặc định (Designing criteria and default
settings)
Tính toàn diện
Tính năng sử dụng
Tốc độ
Xem xét đến lựa chọn thứ 2 khi:
Bạn mới lần đầu thiết kế
Bạn không có nhiều sự tự tin trong lựa chọn kết quả
Lớp Học Phần VNUA - Khoa Nông Học - Học Viện Nông Nghiệp Việt Nam
https://sites.google.com/site/lophocphank57vnua/home


























