
Th c hành bioinformaticsự
(Bài th c hành 1 và 2)ự
L u ý:ư
1. Th c hành t i phòng máy tính c a Khoa Tin h c, t ng 1 nhà B. Th i gian b t đ u tự ạ ủ ọ ầ ờ ắ ầ ừ
8h00 đ n 11h30ế
2. Có 4 bài th c hành (t ng ng v i 4 bu i cho m i nhóm)ự ươ ứ ớ ổ ỗ
3. Đ ngh các nhóm iề ị n tài li u h ng d n th c hành tr c m i bu i th c t pệ ướ ẫ ự ướ ỗ ổ ự ậ
4. Bài th c hành 1 và 2 s đ c ti n hành trong 2 bu i. M i bu i làm 2 ph n.ự ẽ ượ ế ổ ỗ ổ ầ
5. K t thúc m i bu i th c hành, n p ngay câu tr l i t i l p.ế ỗ ổ ự ộ ả ờ ạ ớ
Ph n 1. ầLàm quen v i Genome browserớ
S d ng máy tính k t n i Internet truy c p vào các trang weử ụ ế ố ậ b sau:
i. UCSC genome browser (http://genome.ucsc.edu/)
ii. Ensembl genome browser (http://www.ensembl.org)
iii. NCBI MapViewer (http://www.ncbi.nlm.nih.gov/mapview/)
iv. ECR Browser (http://ecrbrowser.dcode.org)
Câu h iỏ :
1. Sau khi tìm hi u các trang web trên hãy gi i thích th nào là genome browserể ở ả ế ?
2. M c đích c a genome browserụ ủ ?
3. Cho bi t có bao nhiêu genome đã đ c xác đ nh trình t các genome browserế ượ ị ự ở ?
4. S d ng UCử ụ SC genome browser, hãy cho bi t ý nghĩa c aế ủ :
BLAT
Gene Sorter
Insilico PCR
Micribial genomes
5. S d ng Ensembl genome browser, hãy cho bi tử ụ ế :
Có bao nhiêu loài trong b linh tr ng (primates) đã đ c xác đ nh trình tộ ưở ượ ị ự
Có bao nhiêu gene mã hóa cho enzym amylase trong genome ng iườ ?
Các enzym này n m trên NST nàoằ ?
6. S d ng Map Viewer, hãy cho bi t:ử ụ ế
Asperginus niger có bao nhiêu NST?
Genome ti th c a sinh v t này có chi u dài bao nhiêu? (tính theo Kilobase_Kb)ể ủ ậ ề
Đ n th i đi m hi n nay có bao nhiêu gene trong genome ti th c a sinh v t nàyế ờ ể ệ ể ủ ậ
đã đ c xác đ nh?ượ ị
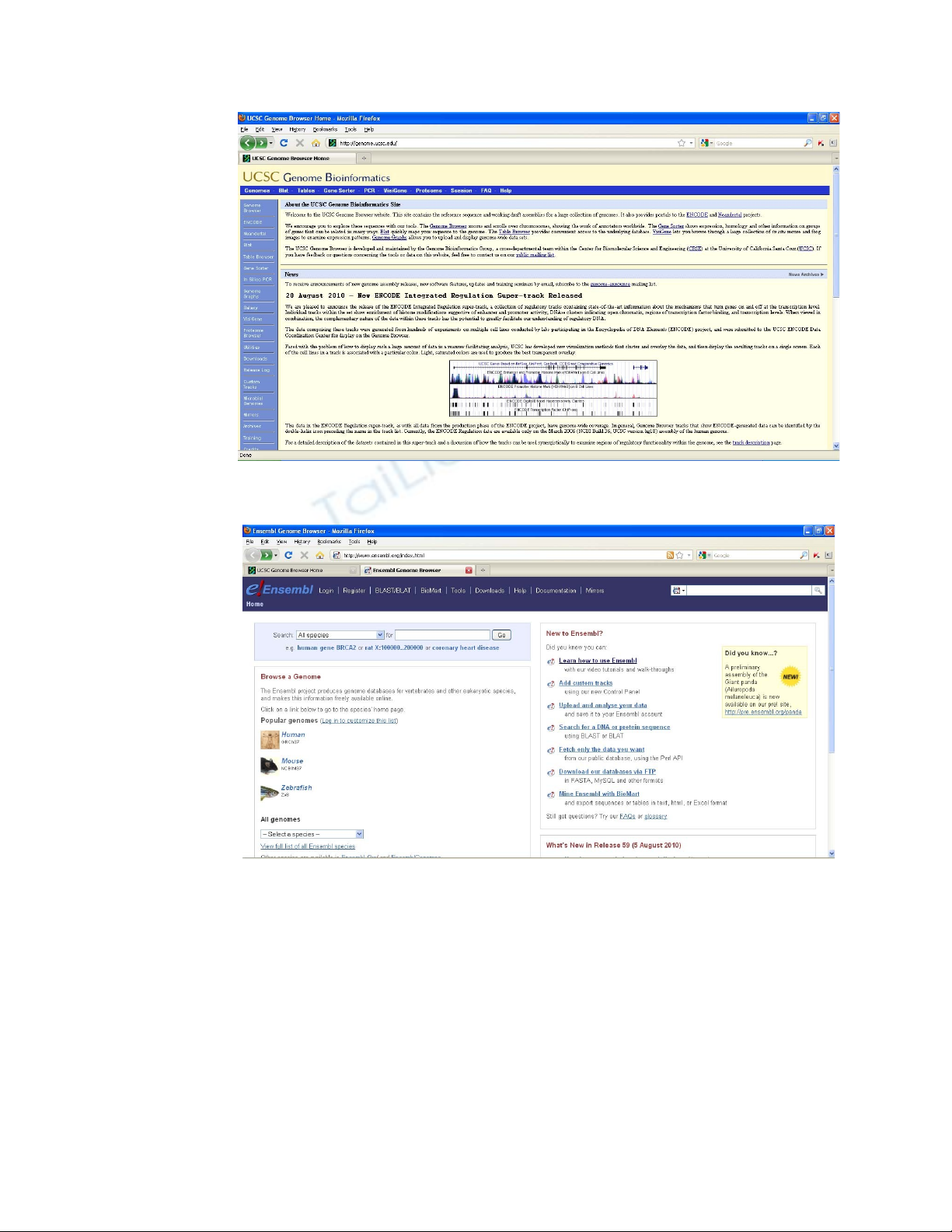
Hình 1. UCSC genome browser
Hình 2. Ensembl genome browser

Ph n 2. ầLàm quen v i c s d li u c a các ngân hàng genớ ơ ở ữ ệ ủ
Truy c p vào các trang web sau:ậ
i. http://www.ncbi.nlm.nih.gov/
ii. http://www.ebi.ac.uk/embl/
iii. http://www.ddbj.nig.ac.jp/
iv. http://www.ebi.ac.uk/uniprot/
v. http://expasy.org/
vi. http://www.pdb.org/pdb/home/home.do
Câu h i:ỏ
1. Hãy cho bi t tên và ý nghĩa c a các trang web trên?ế ủ
2. Truy c p vào các trang web i, ii, iii và iv trên. Hãy k tên nh ng CSDL chính và ý nghĩaậ ở ể ữ
c a chúng.ủ
3. Truy c p vào trang web ậhttp://www.ncbi.nlm.nih.gov/
•Trong m c tìm ki m Search, tìm m c All database. Hãy cho bi t có t t c baoụ ế ụ ế ấ ả
nhiêu CSDL? K tên và nói ng n g n ý nghĩa c a t ng CSDL.ể ắ ọ ủ ừ
•Trong CSDL gene, hãy cho bi t có bao nhiêu gene mã hóa cho enzyme amylase đãế
đ c bi t cho t i nay? ( t t c các loài có trong CSDL)ượ ế ớ ở ấ ả
4. Gene mã hóa cho enzyme alpha amylase ởBacillus subtilis 168 có gene ID là 938356,
locus tag là: BSU03040, s truy c p trong NCBI là AL009126, NCBI Reference Sequenceố ậ
là NC_000964.3, GI:255767013.
Hãy s d ng CSDL c a ngân hàng gene NCBI đ tr l i các câu h i sau:ử ụ ủ ể ả ờ ỏ
•Thông tin tóm t t v gene này? (bao g m gene symbol, gene description, locus tag,ắ ề ồ
gene type, RefSeq status, organism, lineage).
•Kích th c c a gene (coding sequence)?ướ ủ
•Có bao nhiêu amino acid trong phân t enzyme do gene đó mã hóaử
•Gene mã hóa cho amylase trên n m tr c và sau gene nào trên NST?ở ằ ướ
•Gene trên liên quan đ n con đ ng chuy n hóa nào c a vi khu n ở ế ườ ể ủ ẩ B. subtilis?
•Trên c s các k t qu đã tìm đ c, tìm hi u thêm các thông tin đ tr l i cácơ ở ế ả ượ ể ể ả ờ
câu h i:ỏ
i. Kích th c genome c a ướ ủ B. subtilis ?
ii. S d ng công c Graphic đ hi n th v trí c a gene trên NST. Cho bi t vử ụ ụ ể ể ị ị ủ ế ị
trí c a gene đó trên NST? (đi m b t đ u và k t thúc). Tìm hi u các geneủ ể ắ ầ ế ể
lân c n và các kho ng tr ng gi a các gene. N u có th i gian hãy phân tíchậ ả ố ữ ế ờ
trình t upstream (đ u 5’) c a gene này.ự ầ ủ
iii. Copy trình t coding sequence (CDS) và trình t amino acid c a gene nàyự ự ủ
d i d ng FASTA và l u l i trên máy tính cho các bài t p ti p theo.ướ ạ ư ạ ậ ế
Ph n 3. Làm quen v i các công c phân tích CSDLầ ớ ụ
Truy c p vào các trang web sau:ậ
vii. http://www.ncbi.nlm.nih.gov/
viii. http://www.ebi.ac.uk/embl/

ix. http://www.ddbj.nig.ac.jp/
x. http://www.ebi.ac.uk/uniprot/
xi. http://expasy.org/
xii. http://www.pdb.org/pdb/home/home.do
Câu h i:ỏ
1. Cho k tên m t s công c phân tích c b n có trong các trang web trên. Nêu ý nghĩa c aể ộ ố ụ ơ ả ủ
chúng?
2. Truy c p vào trang web (EMBL) http://www.ebi.ac.uk/embl/ậ
•Hãy quan sát các công c phân tích có trong m c Tools. K tên và nói ng n g n ýụ ụ ể ắ ọ
nghĩa c a m t s công c phân tích c b n (ít nh t 5 nhóm công c )ủ ộ ố ụ ơ ả ấ ụ
•V n trong ph n Tools c a EMBL, tìm hi u nhóm công c phân tích Similarity &ẫ ầ ủ ể ụ
Homology. Hãy cho bi t ý nghĩa và m c đích s d ng c a các công c sau:ế ụ ử ụ ủ ụ
i. BLAST2-WU Protein
ii. BLAST2-WU Nucleotide
iii. BLAST2-NCBI
iv. BLAST2-NCBI Nucleotide
v. BLAST2-NCBI EVEC
vi. PSI-BLAST (old version)
vii. PSI-BLAST (new version)
viii. PHI-BLAST
ix. FASTA Nucleotide
x. FASTA Protein
xi. FASTA-Proteome Server
xii. FASTA-Genome Server
xiii. FASTA-WGS Server
3. Truy c p vào trang web c a NCBI theo đ a ch sau: ậ ủ ị ỉ http://www.ncbi.nlm.nih.gov/Tools/
Hãy li t kê các công c phân tích và nêu ý nghĩa c a chúngệ ụ ủ
4. Truy c p vào trang web (ExPASy) http://expasy.org/ậ
Trong ph n Tools and Software, ch n m c ExPASy Proteomics tools. Hãy choầ ọ ụ
bi t có bao nhiêu nhóm công c phân tích proteomic? Trong m i nhóm công c ,ế ụ ỗ ụ
hãy k ra ít nh t 2 công c phân tích và nêu ý nghĩa c a chúng?ể ấ ụ ủ
Ph n 4. Làm quen v i vi c phân tích CSDL trình t sinh h c ầ ớ ệ ự ọ
4.1. Phân tích b n đ gi i h nả ồ ớ ạ
Truy c p vào các đ a ch sau:ậ ị ỉ
Tên trang web Đ a chị ỉ
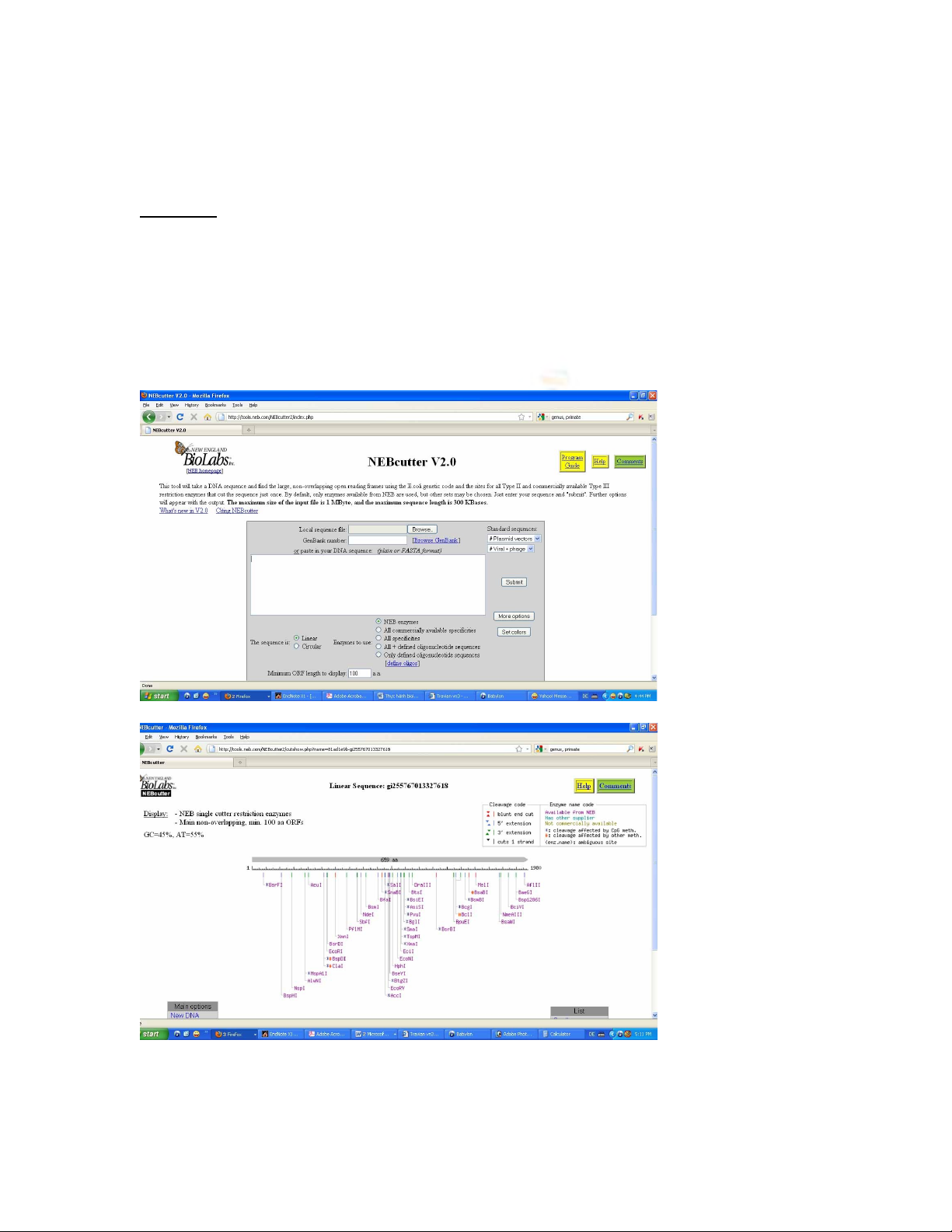
NEB Cutter http://tools.neb.com/NEBcutter2/index.php
Restriction/Mapper http://arbl.cvmbs.colostate.edu/molkit/mapper/index.html
http://www.restrictionmapper.org/
Webcutter http://www.ccsi.com/firstmarket/cutter/cut2.html
In Silico Restriction insilico.ehu.es
WatCut http://watcut.uwaterloo.ca/watcut/watcut/
EnzymeX http://mekentosj.com/enzymex/
Silent http://bioweb.pasteur.fr/seqanal/interfaces/silent.html
Câu h iỏ :
1. Hãy cho bi t ý nghĩa c a các trang web trênế ủ
2. Truy c p vào trang web NEB Cutter. Hãy paste trình t c a gene mã hóa cho enzym amylaseậ ự ủ
c a ủB. subtilis bài t p trên vào ô tr ng ho c đi n đ a ch ID c a gene. Quan sát b n đ c aở ậ ố ặ ề ị ỉ ủ ả ồ ủ
các enzym c t gi i h n đ i v i gene trên.ắ ớ ạ ố ớ
3. Áp d ng t ng t v i các công c trong các trang web còn l i. Cho bi t website nào cungụ ươ ự ớ ụ ạ ế
c p công c phân tích b n đ gi i h n cho k t qu t t nh t (d thao tác, thu n ti n, k tấ ụ ả ồ ớ ạ ế ả ố ấ ễ ậ ệ ế
qu hi n th rõ ràng).ả ể ị
Hình 3. K t qu phân tích b n đ gi i h n c a enzym alpha amylase c a ế ả ả ồ ớ ạ ủ ủ B. subtilis





![Câu hỏi ôn tập hóa sinh thực phẩm [chuẩn nhất]](https://cdn.tailieu.vn/images/document/thumbnail/2018/20181019/ayunn123/135x160/2711539952639.jpg)
![Ôn tập thực hành môn Thực vật [chuẩn nhất]](https://cdn.tailieu.vn/images/document/thumbnail/2015/20151028/ngochunghcm/135x160/1692190693.jpg)

![Định lượng Vitamin C: Bài 8 [Hướng Dẫn Chi Tiết]](https://cdn.tailieu.vn/images/document/thumbnail/2014/20140222/minhtriet16/135x160/7291393079428.jpg)




![Bài giảng Giáp xác chân mái chèo [mới nhất]](https://cdn.tailieu.vn/images/document/thumbnail/2025/20250927/lethihongthuy2402@gmail.com/135x160/92891759114976.jpg)



![Tài liệu học tập Chuyên đề tế bào [mới nhất]](https://cdn.tailieu.vn/images/document/thumbnail/2025/20250906/huutuan0/135x160/56151757299182.jpg)






