
Tuan V. Nguyen
Senior Principal Research Fellow, Garvan Institute of Medical Research
Professor, UNSW School of Public Health and Community Medicine
Professor of Predictive Medicine, University of Technology Sydney
Adj. Professor of Epidemiology and Biostatistics,
School of Medicine Sydney, University of Notre Dame Australia
Phân tích dữ liệu và ứng dụng | Đại học Dược Hà Nội | 12/6 to 17/6/2019 © Tuan V. Nguyen

Nội dung
•Mô hình hồi qui tuyến tính đa biến
•Ứng dụng 1: đánh giá mối liên quan (association / assessment)
•Ứng dụng 2: hiệu chỉnh cho yếu tố nhiễu (adjustment)
•Ứng dụng 3: mô hình tiên lượng (prediction)

Xây dựng mô hình tiên lượng tỉ trọng mỡ
• Để đo tỉ trọng mỡ (pcfat), cần phải có máy DXA (đắt tiền)
•Có thể xây dựng một mô hình tiên lượng pcfat chỉ cần dùng các yếu tố
'thường qui' (có thể thu thập từ bệnh nhân)
•Các biến thuờng qui: giới tính (gender), chiều cao (height), cân nặng
(weight), tỉ trọng cơ thể (bmi), tuổi (age)
•Giải pháp: mô hình hồi qui tuyến tính

Xây dựng mô hình tiên lượng tỉ trọng mỡ
•Bước 1: Phân tích khai thác (exploratory analysis)
•Bước 2: Tìm các biến liên quan (có giá trị thống kê)
•Bước 3: Chia dữ liệu thành 2 nhóm: development và validation
•Bước 4: Phát triển mô hình dựa vào biến bước 2 trên nhóm development
•Bước 5: Kiểm tra mô hình ở bước 5 trên nhóm validation
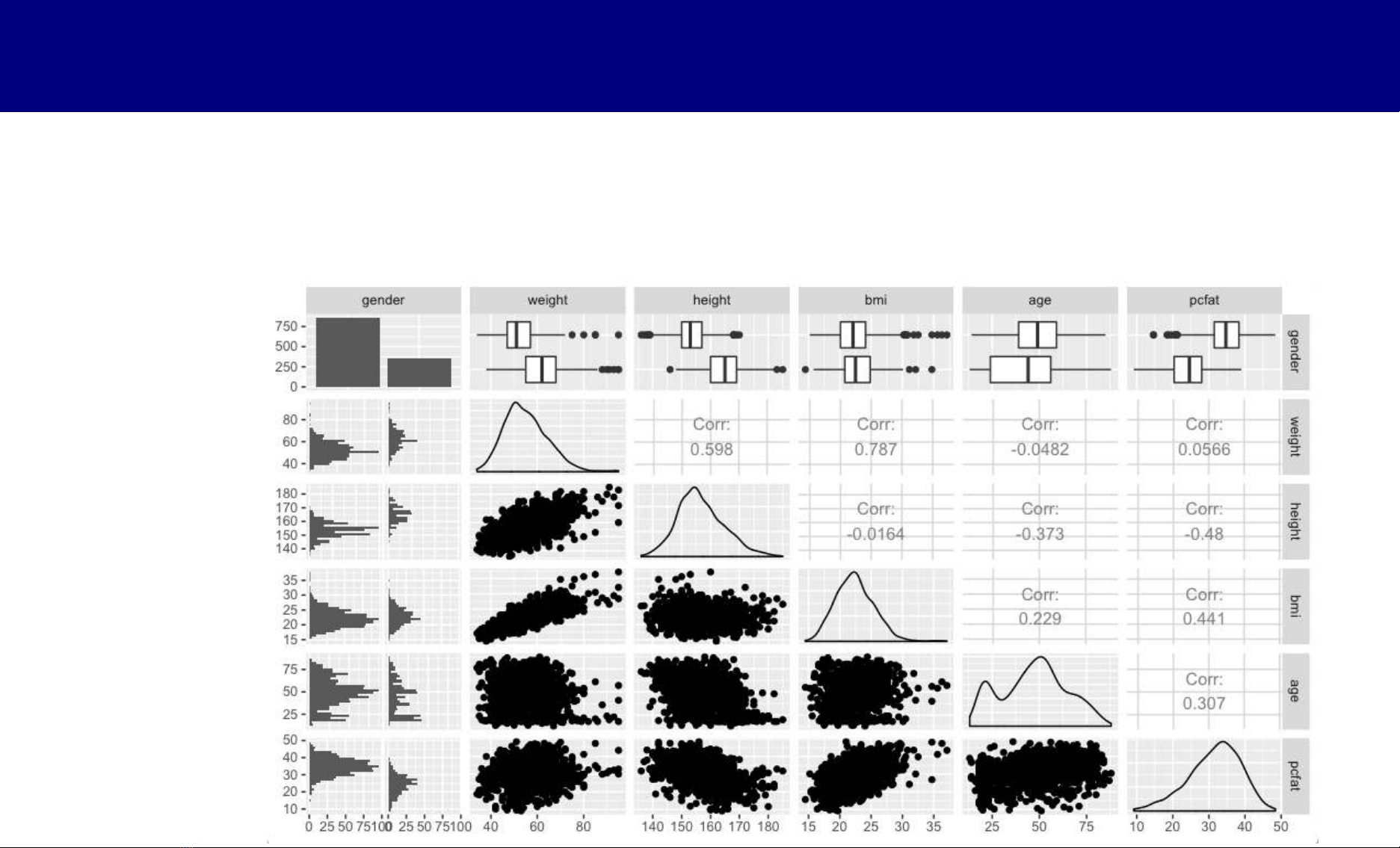
Bước 1: Phân tích mô tả / khai thác
# Các biến có thể liên quan
dat = ob[, c("gender", "weight", "height", "bmi", "age", "pcfat")]
library(GGally)
ggpairs(dat)



![Bài giảng Hồi quy tuyến tính đa biến [chuẩn SEO]](https://cdn.tailieu.vn/images/document/thumbnail/2021/20210804/toan5ks1/135x160/5551628051569.jpg)













![Bài tập Đại số tuyến tính [chuẩn nhất]](https://cdn.tailieu.vn/images/document/thumbnail/2025/20250930/dkieu2177@gmail.com/135x160/79831759288818.jpg)








